SCI论文(www.lunwensci.com):
摘要:目的应用生物信息学分析miR-194-5p序列,预测其潜在的靶基因及信号通路。方法通过PubMed、NCBI、UCSC等在线数据库查找miR-194-5p碱基序列,借助miRanda、TargetScan和miRDB预测miR-194-5p作用靶点,应用STRING平台和Cytoscape 3.7.0软件构建蛋白互作网络图,运用David在线数据库进行GeneOntology(GO)分析和KEGG信号转导通路富集分析。结果已知的成熟miR-194-5p序列在不同物种间高度保守。蛋白质互作分析显示,miR-194-5p预测的靶基因所编码蛋白质间存在复杂的相互作用,尤其是靶基因HIST1H2BD、CASK、CHD4、CUL4B、DYRK1A等编码的蛋白质起关键作用。GO分析发现其靶基因参与Golgi组织、伤口愈合、细胞对DNA损伤刺激的反应等生物学过程,KEGG分析显示富集在泛素介导的蛋白水解信号通路。结论分析结果初步提示miR-194-5p通过调控靶基因广泛参与多种重要的生物学过程,为后期开展miR-194-5p实验研究提供新的思路与线索。
关键词:miR-194-5p;生物信息学;靶基因;信号通路
本文引用格式:谢香莲,涂兴明,汪悦东,等.miR-194-5p靶基因预测及信号通路的生物信息学分析[J].世界最新医学信息文摘,2019,19(59):20-22.
Bioinformatics Analysis of MiR-194-5p Target Gene Prediction and Signaling Pathways
XIE Xiang-lian1,TU Xing-ming2,WANG Yue-dong3,ZHU Gen-fu2*
(Guangzhou liwan district hailong street community health service center,Guangzhou Guangdong;2.The third affiliated hospital of guangzhou university of traditional Chinese medicine,Guagnzhou Guangdong;3.Guangzhou university of traditional Chinese medicine,Guagnzhou Guangdong)
ABSTRACT:Objective The miR-194-5p sequence was analyzed using bioinformatics and predict its potential target genes and signaling pathways.Methods The miR-194-5p base sequence was searched by onlinedatabases such as PubMed,NCBI and UCSC.The target of miR-194-5p was predicted by miRanda,TargetScan and miRDB,and the protein interaction network was constructed by STRING platform and Cytoscape 3.7.0 software.Gene Ontology(GO)analysis and KEGG signal transduction pathway enrichment analysis were performed using David's online database.Results The known mature miR-194-5p sequencesare highly conserved among different species.Protein interaction analysis showed that miR-194-5p predicted complex interactions between the proteins encoded by the target genes,especially the proteins encoded by the target genes HIST1H2BD,CASK,CHD4,CUL4B,DYRK1A and so on.GO analysis revealed that the target gene is involved in biological processes such as Golgi tissue,wound healing,and cell response to DNA damage stimulation.KEGG analysis showed enrichment in the ubiquitin-mediated proteolytic signaling pathway.Conclusion The results suggest that miR-194-5p can participate in a variety of important biological processes by regulating target genes,which provides new ideas and clues for the future research of miR-194-5p.
KEY WORDS:miR-194-5p;Bioinformatics;Target gene;Signal pathway
0引言
miRNAs(microRNAs)存在于植物、动物和病毒中,充当大多数生理和病理细胞过程的调节剂,其长度为19至23个核糖核苷酸(nt),通过特异性结合和切割信使RNA(mRNA),主要作用于靶基因mRNA的3'非翻译区(3'UTR),抑制翻译和使mRNA尾部去腺苷化来抑制其同源靶基因的表达。miRNAs作为一类新的基因调控因子,通过直接或间接调控靶基因表达,参与调控众多生物学过程和疾病的发生过程,miRNA对细胞增殖、死亡、免疫、神经系统模式等生物学过程的调控至关重要[1-2]等。人miR-194(hsa-miR-194)的编码基因位于1、11号染色体的长臂上。多项研究表明,miR-194在肝细胞癌、结肠癌、肾细胞癌、子宫内膜癌等恶性肿瘤中异常表达,提示其在肿瘤的发生和进展中起重要作用。本研究通过生物学分析,预测miR-194-5p的靶基因,并对其靶基因集合进行蛋白质互作网络分析、GO功能富集分析和KEGG通路分析,为开展miR-194-5p的功能探讨、机制研究提供理论基础。
1材料与方法
1.1miR-194-5p基本信息查找
使用PubMed查找已发表的miR-194研究文献;利用UCSC、miRBase、NCBI等在线数据库查找miR-194-5p的碱基序列、物种保守性、染色体定位等基本信息。
1.2miR-194-5p靶基因预测
运用在线靶基因预测软件miRanda、TargetScan和miRDB预测miR-194-5p的靶基因,为了降低假阳性率,取三者的交集基因组成进行下一步分析,Venny2.1.0画韦恩图显示交集结果。
1.3PPI网络构建
首先将所得的靶基因集合导入String10.5在线数据库进行互作预测分析并导出互作关系的tsv文件,然后再将得到的分析结果导入Cytoscape_v3.7.0获得靶基因集合编码蛋白质间相互作用(protein-proteininteraction,PPI)网络图,利用CytoHubba插件筛选兴趣靶基因。
1.4GO和KEGG分析
根据Gene ontology数据库查找的基因GO注释,以本物种的所有基因为背景基因,用超几何分步法计算P值,显著性阈值取P<0.05,获得相对于背景基因具有统计意义的GO高频率注释。Geneontology功能注释由细胞组件、分子功能、生物过程3个部分组成。用DAVID数据库对miR-194-5p预测靶基因集合进行生物通路富集分析。通过Fisher’sexact test计算P值,以KEGG通路富集程度的P<0.05为显著性阈值,获得相对于背景具有统计学意义的基因集合信号转导通路。
2结果与分析
2.1miR-194-5p参与的疾病
PubMed文献检索发现,miR-194-5p主要参与肿瘤的发生,包括肝细胞癌、非小细胞肺癌、结肠癌、肾细胞癌;miR-194-5p主要作为肝上皮细胞、绝经后骨质疏松症、骨髓增生异常综合征的标志物;另外,miR-194-5p参与软骨形成分化、促进成骨细胞分化诱导细胞周期停滞等过程。
2.2miR-194-5p定位及序列保守性分析
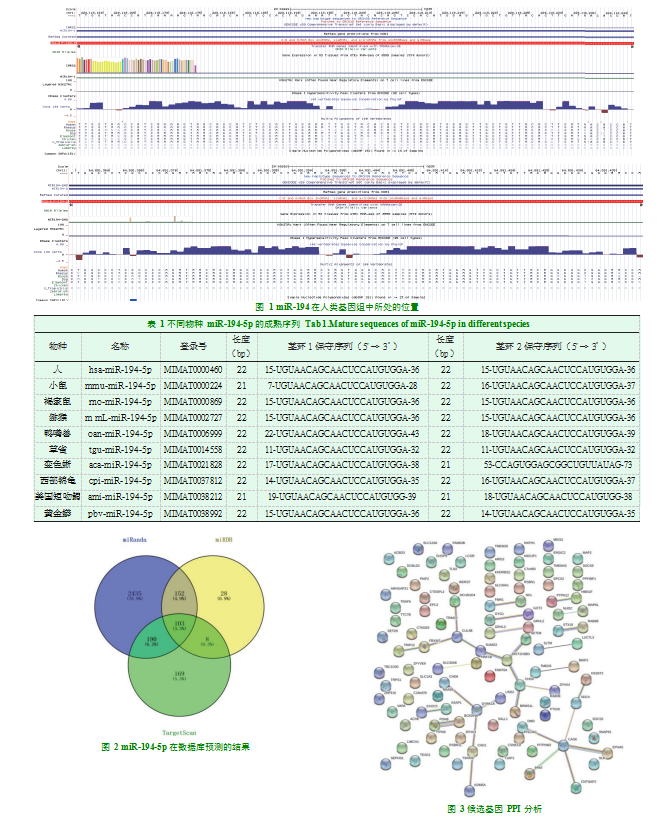
在线检索miRBase、NCBI、UCSC数据库发现人(hsa)miR-194-5p基因序列号为MIMAT0000460,茎环1定位于1q41染色体区域中的一个簇内(图1),其基因位点chr1:220118157-220118241;茎环2定位于11q13,1染色体区域中的一个簇内,其基因位点chr11:64891355-64891439。通过miRBase对不同物种的miR-194-5p序列进行比对分析,发现miR-194-5p的成熟序列“UGUAACAGCAACUCCAUGUGGA”在小鼠、褐家鼠、猕猴、鸭嘴兽、草雀等多物种间高度保守(表1)。
2.3miR-194-5p的靶基因预测
选用3个在线数据库miRanda、TargetScan和miRDB分别预测出2880、470和291个靶基因,合并三个在线数据库的结果得到103个靶基因(图2)。
2.4靶基因编码蛋白质间的相互作用预测
将3个数据库预测到的靶基因取交集后,导入String10.5数据库在线分析靶基因编码蛋白质间的相互作用,然后再将String分析结果导入Cytoscape_v3.7.0软件绘制PPI图。结果显示,miR-194-5p的靶基因编码蛋白质间具有复杂的相互作用,尤其是靶基因HIST1H2BD、CASK、CHD4、CUL4B、DYRK1A等编码蛋白质在相互作用网络图中发挥稳定结构的作用(图3、图4)。
2.5靶基因GO分析
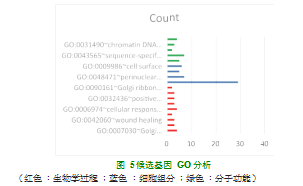
对3个数据库都预测到的103靶基因做GO功能注释,发现miR-194-5p靶基因的GO生物学过程富集在Golgi组织、伤口愈合、细胞对DNA损伤刺激的反应、昼夜调节基因表达等,细胞成分富集在细胞核、细胞质的核周区域、Golgi膜、细胞表面,分子功能富集在泛素蛋白连接酶结合、序列特异性DNA结合、相同的蛋白结合、泛素介导的蛋白水解等(图5)。
2.6靶基因KEGG分析
对3个数据库都预测到的103个靶基因进行KEGG信号转导通路富集性分析,显示富集在泛素介导的蛋白水解信号通路。
3讨论
miRNA是一类小的内源性非编码基因,在转录后调控以及其他重要的生物学过程中起重要作用。越来越多的证据表明,miRNA广泛参与癌症的病理学研究,miRNA的发现为肿瘤的发病机制提供了新的思路,也为肿瘤的诊断与治疗提供了新的策略[3-5]。miRanda是依据miRNA与靶基因3'UTR的匹配、miRNA和mRNA双链间的热稳定性以及保守性3个方面进行靶基因预测[6]。miRDB在线数据库包括由6709个miRNA调节的210万个预测基因靶标,采用NCBI RefSeq数据库来鉴定3’-UTR序列,使用MirTarget算法进行目标预测[7]。TargetScan预测软件通过RNA与RNA之间相互作用的热力学模型与序列比值对分析相互结合预测不同物种间保守miRNA的靶标。
Cullin 4B(CUL4B)是一种组装CRL4B泛素连接酶复合物的支架蛋白,在许多类型的癌症中过表达,并通过表观遗传机制抑制许多肿瘤抑制因子。Yuan J等[8]发现CUL4B可以通过转录抑制Wnt拮抗剂来上调人HCC中的Wnt/β-连环蛋白信号传导,从而导致肝细胞癌。Gyg1,Setd5,SUMO2,Cln4b和RAP2B是的miR-194的靶点,而EREG,ALCAM,和的MSN是miR-192靶点,这些发现揭示了由miR-194/192控制的新型HNF4α网络,其可通过抑制参与去分化和肿瘤发生的基因的表达而在维持肝细胞分化状态中起关键作用,有助于早期HCC检测的诊断标志物的开发[9]。沉默CUL4B基因表达可有效抑制骨肉瘤细胞增殖并诱导细胞凋亡[10]。Jun等[11]发现microRNA-194和CUL4B蛋白在非小细胞肺癌症标本中呈负相关,并证明miR-194可通过直接靶向其3’-UTR而下调CUL4B,microRNA-194和CUL4B蛋白在非小细胞肺癌症标本中呈负相关,并证明miR-194可通过直接靶向其3’-UTR而下调CUL4B。Fbxw7是F-box蛋白质家族成员,它是SCF型泛素连接酶复合体中负责底物识别的部分。荧光素酶报告基因证实含有F-box/WD重复序列的蛋白7(Fbxw-7)可能是miR-194的直接靶标。miR-194的表达通过靶向Fbxw-7促进乳腺癌细胞的增殖,并可作为乳腺癌治疗的生物标志物和新靶点[12]。荧光素酶报告基因证实miR-194显着降低染色质结构域解旋酶DNA结合蛋白1(CHD1)3’UTR的活性[13]。
综上所示,本研究通过生物信息学方法得到miR-194的序列分析图、作用靶基因、GO分析和KEGG信号通路分析、靶基因蛋白互作网等结果,为进一步探索miR-194在疾病的发生、发展及分子机制等方面提供理论指导。
参考文献
[1]Baba O K,Ali A,Shafi M,et al.MicroRNAs as Early Detection Biomarker in Cancer[J].Int.J.Curr.Microbiol.App.Sci,2017,6(11):5419-5424.
[2]Vidigal J A,Ventura A.The biological functions of miRNAs:lessons from in vivo studies[J].Trends in cell biology,2015,25(3):137-147.
[3]Lo Russo G,Tessari A,Capece M,et al.MicroRNAs for the Diagnosis and Management of Malignant Pleural Mesothelioma:A Literature Review[J].Frontiers in oncology,2018,8:650.
[4]Zhang Z,Shao L,Wang Y.MicroRNA-501-3p restricts prostate cancer growth through regulating cell cycle-related and expression-elevated protein in tumor/cyclin D1 signaling[J].Biochemical and biophysical research communications,2019.
[5]Siriwardhana C,Khadka VS,Chen JJ.Development of a miRNA-seq based prognostic signature in lung adenocarcinoma[J].BMC cancer,2019,19(1):34.
[6]Huang Y,Zou Q,Song H,et al.A study of miRNAs targets prediction and experimental validation[J].Protein&cell,2010,1(11):979-986.
[7]Wong N,Wang X.miRDB:an online resource for microRNA target prediction and functional annotations[J].Nucleic acids research,2014,43(D1):D146-D152.
[8]Yuan J,Han B,Hu H,et al.CUL4B activates Wnt/β‐catenin signalling in hepatocellular carcinoma by repressing Wnt antagonists[J].The Journal of pathology,2015,235(5):784-795.
[9]Morimoto A,Kannari M,Tsuchida Y,et al.An HNF4α-microRNA-194/192 Signaling Axis Maintains Hepatic Cell Function[J].Journal of Biological Chemistry,2017:jbc.M117.785592.
[10]Chen Z,Shen B L,Fu Q G,et al.CUL4B promotes proliferation and inhibits apoptosis of human osteosarcoma cells[J].Oncology reports,2014,32(5):2047-2053.
[11]Mi J,Zou Y,Lin X,et al.Dysregulation of th e miR‐194-CUL4B negative feedback loop drives tumorigenesis in non‐smal‐l cell lung carcinoma[J].Molecular oncology,2017,11(3):305-319.
[12]Chen Y,Wei H,Liu Y,et al.Promotional effect of microRNA-194 on breast cancer cells via targeting F-box/WD repeat-containing protein 7[J].Oncology letters,2018,15(4):4439-4444.
[13]Lu H,Lei X,Liu J,et al.Regulation of hepatic microRNA expression by hepatocyte nuclear factor 4 alpha[J].World journal of hepatology,2017,9(4):191.
关注SCI论文创作发表,寻求SCI论文修改润色、SCI论文代发表等服务支撑,请锁定SCI论文网! 文章出自SCI论文网转载请注明出处:https://www.lunwensci.com/yixuelunwen/16625.html