SCI论文(www.lunwensci.com):
摘要:目的探讨应用TCGA数据库筛选胰腺癌患者差异表达的miRNAs,运用生物信息学预测其相关靶基因。方法应用R语言筛选与正常胰腺组织相比差异表达的miRNAs,TCGA数据库下载胰腺癌miRNA_HiSeq_gene资料包,运用Microsoft Office Excel软件对数据包分析处理,应用GraphPad Prism 6绘制胰腺癌患者miRNAs差异表达、临床分期,Kaplan-Meier生存曲线,使用生物信息学网站预测其相关靶基因。结果筛选出hsa-miR-192-5p、hsa-miR-203a-3p和hsa-miR-451a在胰腺癌患者有明显差异表达(P<0.05),MCM4、REV3L、ZBTB4为hsa-miR-192-5p的靶基因,ZNF148、TMEM69为hsa-miR-203a-3p的靶基因,EREG为hsa-miR-451a靶基因。结论差异表达的miRNAs和靶基因对胰腺癌的诊断和预后有重要影响,可以作为肿瘤生物标志物对其进行进一步研究,对癌症前期预防及诊疗提供了有力的依据。
关键词:TCGA;差异表达miRNAs;靶基因预测;数据筛选
本文引用格式:王源,苏静慧,侯晓丽,等.运用TCGA分析胰腺癌患者差异表达miRNAs及其靶基因预测[J].世界最新医学信息文摘,2019,19(71):38-43.
Analysis of Differentially Expressed miRNAs and their Target Genes in Pancreatic Cancer Patients by TCGA Database
WANG Yuan1,SU Jing-hui1,HOU Xiao-li2,ZHANG Rong-hua2,WANG Mei-mei2,XIONG Ya-nan2,ZHANG Guang-ling2*
(1.North China University of Technology,Grade 17 of Graduate School,Tangshan Hebei;2.Basic Medical College of North China University of Technology,Hebei Key Laboratory of Chronic Diseases,Tangshan Key Laboratory of Clinical Basic Research on Chronic Diseases,Tangshan Hebei)
ABSTRACT:Objective To investigate the differential expression of miRNAs in patients with pancreatic cancer using the TCGA database and to predict target genes using bioinformatics sites.Methods R language was used to screen differentially expressed miRNAs compared with normal pancreatic tissue.TCGA database downloaded pancreatic cancer miRNA_HiSeq_gene data package,using Microsoft Office Excel software to analyze data packets,and GraphPad Prism 6 was used to map differential expression and clinical stage,Kaplan-Meier survival curves of miRNAs in patients with pancreatic cancer.Using bioinformatics sites to predict their relevant target genes.Results Hsa-miR-192-5p,hsa-miR-203a-3p and hsa-miR-451a were screened in patients with pancreatic cancer(P<0.05).MCM4,REV3L and ZBTB4 were the target genes of hsa-miR-192-5p.ZNF148 and TMEM69 are the target genes of hsa-miR-203a-3p,and EREG is the target gene of hsa-miR-451a.Conclusion Differentially expressed miRNAs and target genes have important effects on the diagnosis and prognosis of pancreatic cancer.They can be further studied as tumor biomarkers,providing a powerful basis for precancerous prevention and treatment.
KEY WORDS:TCGA;Differentially expressed miRNAs;Target gene prediction;Data screening
0引言
肿瘤基因组图谱(TCGA)由美国癌症研究所(NCI)和美国人类基因研究所(NHGRI)于2005年联合启动的项目,它的诞生为科学研究提供原始临床数据,利用基因组平台分析了30多种肿瘤或癌症类型[1],包含11000个临床肿瘤样本遗传基因、蛋白质组学、表观遗传进行了测序工作,覆盖了半数癌症临床病例的基因突变[2]。胰腺癌(pancreatic cancer,PC)的发病率近年来呈上升趋势。是胃肠道肿瘤患者死亡的第二位原因,也是美国肿瘤死亡第四大因素[3]。是预后最差的恶性肿瘤之一,总体5年生存率不到5%,故被称为“癌中之王”[4]。本研究旨在通过TCGA分析与胰腺癌发生有关miRNAs,预测相关靶基因,从而为胰腺癌的治疗提供新的分子靶点。
MicroRNA(miRNA)是一类短链非编码RNA,通过与其靶基因的3’-非编码区(3’-UTR)相互作用调节靶基因的表达,导致靶基因翻译抑制或降解[5]。不同miRNA在不同癌症中的调节机制不同,作为最有效的调控分子之一,影响人类遗传学的改变,例如染色体重排,基因组扩增,缺失或点突变等,通过区域内miRNA表达水平变化,调控下游基因表达,在癌症进程中发挥关键作用[6]。研究表明,胰腺癌中miR-27a,miR-27b,miR-9,miR10a和miR-10b异常表达[7],近期的研究表明特定miRNA的突变或错误表达与各种人类癌症息息相关,miRNA可作为肿瘤抑制因子或促癌基因直接或间接调节靶基因的表达,在肿瘤细胞的侵袭,转移和化学抗性中起关键作用。已有研究证实,胰腺癌中异常表达的miRNAs,能够成为胰腺癌治疗的新靶点,在临床治疗中具有积极效果[8]。
1方法
1.1差异表达miRNAs的筛选
登录Google Chrome浏览器,输入网址:https://xenabrowser.net/datapages/,登录TCGA数据库,查找胰腺癌相关数据包进行下载,使用pheatmap程序包分析,对差异miRNAs采用R语言edgeR软件包提取,挑选正常胰腺组织(normal Mean)与胰腺癌患者组织(Tumor Mean)差异倍数(Fold Change)大于2倍,即logFC>2,且P<0.05,作为差异表达有统计学意义的miRNAs。
1.2分析差异表达miRNAs保守性
对筛选出的hsa-miR-192-5p,hsa-miR-203a-3p和hsa-miR-451a进行保守性分析,使用miRBase和TargetScan网站查询其保守性,发现高表达组中hsa-miR-192-5p、hsa-miR-203a-3p基因序列具有物种保守性,选为差异表达的miRNAs。低表达组中hsa-miR-451a基因序列具有物种保守性,选为胰腺癌组织中差异表达的miRNAs。
1.3分析差异表达miRNAs患者的生存曲线,临床分期
利用Kaplan-Meier分析胰腺癌组织中低表达组与高表达组患者的总体生存率,进一步采用Graphpad6.0制作Kaplan-Meier生存分析图。TCGA分析出基因表达数据和临床数据相结合,找到肿瘤分期诊断信息,制作差异表达的miRNA箱式图与TNM分期散点图(stageⅰ、stageⅱ、stageⅲ、stageⅳ)。患者生存曲线具有统计学意义(P<0.05)的miRNAs进一步分析它们在胰腺癌组织中的差异表达和临床分期。
1.4相关靶基因预测
利用DIANA TOOLS v5.0生物信息网站预测高表达组差异显著的hsa-miR-192-5p、hsa-miR-203a-3p和低表达组差异显著的hsa-miR-451a的靶基因,并按与靶基因结合程度评分,筛选具有较高评分排名的靶基因,每个miRNA至少预测10个靶基因。
1.5分析靶基因生存曲线及其与miRNAs相关性分析
接下来运用GEPIA数据库分析靶基因在胰腺癌中的生存曲线,排除已经验证过的靶基因和无原始数据的靶基因,对具有统计学意义的靶基因(P<0.05)继续分析它们与显著表达miRNAs之间的相关性。
2结果
2.1TCGA数据库筛选差异表达miRNAs
与正常胰腺组织相比较,胰腺癌患者组织中高表达组miRNAs 9个,低表达组miRNAs 9个,差异表达均有统计学意义(P<0.05)(表1-2)。
2.2差异表达miRNAs物种保守性预测结果
高表达组hsa-miR-192-5p、hsa-miR-194-5p、hsa-miR-203a-3p、hsa-miR-203a-5p、hsa-miR-592基因序列具有广泛物种保守性(图1);低表达组hsa-miR-142-5p、hsa-miR-150-5p、hsa-miR-206、hsa-miR-451a基因序列具有物种保守性(图2)。
2.3物种保守性差异表达miRNAs生存曲线
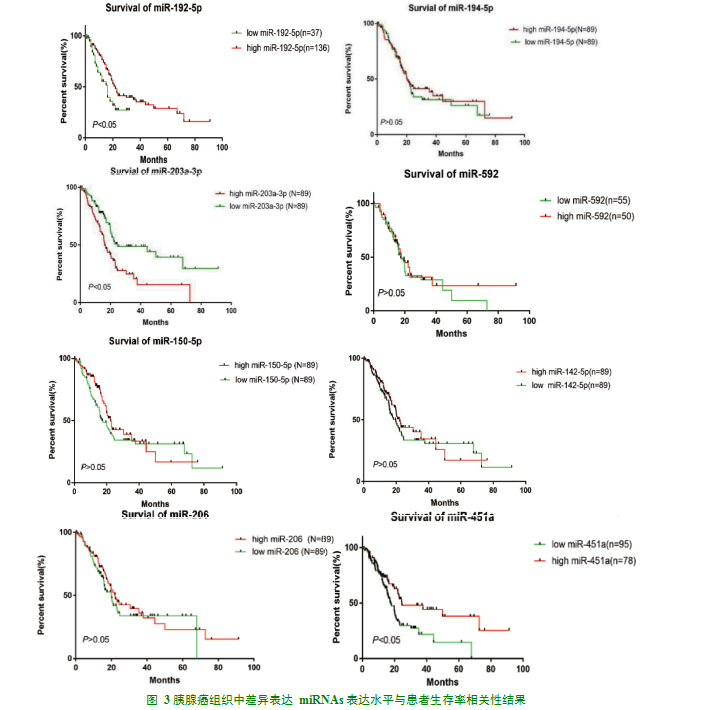
分析胰腺癌组织中差异表达并且物种保守的miRNAs,其表达水平与患者生存相关,结果提示胰腺癌患者组织中高表达组hsa-miR-192-5p、hsa-miR-203a-3p及低表达组hsa-miR-451a的表达水平与患者生存率相关,结果具有统计学意义(P<0.05)(图3)。
胰腺癌患者高表达组生存率具有统计学意义(P<0.05)的miRNAs有hsa-miR-192-5p、hsa-miR-203a-3p。低表达组患者生存率具有统计意义(P<0.05)的miRNAs有hsa-miR-451a进一步分析它们在胰腺癌组织中临床分期(图4)。
2.4靶基因结果的预测
利用DIANA TOOLS v5.0生物信息网站预测高表达组差异显著的hsa-miR-192-5p、hsa-miR-203a-3p和低表达组差异显著hsa-miR-451a的靶基因,并按与靶基因结合程度评分,筛选具有较高评分的靶基因(图5)。
2.5筛选预测靶基因的生存曲线,分析miRNAs与靶基因的相关
miR-192-5p候选靶基因与胰腺癌患者生存相关性分析,排除已经验证的靶基因,结果提示表达水平与患者生存相关且具有统计学意义(P<0.05)的候选靶基因为MCM4、REV3L、ZBTB4,对有统计学意义(P<0.05)的靶基因继续分析它们与miR-192-5p的相关性。MCM4、REV3L、ZBTB4分别与miR-192-5p负相关(R值分别为-0.098、-0.217、-0.333)(图6)。
接下来分析miR-203a-3p候选靶基因与胰腺癌患者生存相关性,结果提示表达水平与患者生存相关且具有统计学意义(P<0.05)的候选靶基因为ZNF148、TMEM69,二者分别与miR-203a-3p正相关(R值分别为0.07115、0.1145)。(图7)。
最后分析miR-451a候选靶基因与胰腺癌患者生存相关性,结果提示表达水平与患者生存相关且具有统计学意义(P<0.05)的候选靶基因为EREG,其与miR-451a是负相关(R值为-0.010)(图8)。
3讨论
miRNA在肿瘤细胞迁移,侵袭,克隆中发挥关键角色,具有促癌或者抑癌的作用,这些功能的最终实现依赖于miRNA对靶基因的分子调控机制[9]。随着生物信息学不断进步和大数据的广泛应用,癌症靶向治疗的关miRNAs作用愈发明确。The Cancer Genome Atlas(TCGA)数据库收录美国癌症患者临床样本及基因相关信息,提供有价值的数据并且免费共享。我们可以从中挖掘和利用这些信息,将统计学和生物信息学知识综合运用到癌症患者的数据分析中,积极发挥其预测作用。
基因疗法在癌症治疗中有着光明前景,寻找胰腺癌有关的miRNAs和靶基因将从分子生物学角度揭示人类疾病发生、发展机制,从而提高疾病预防的认知水平,丰富治疗手段,降低恶性肿瘤的病死率,为全人类健康造福。TCGA汇集了全球范围内高通量测序、基因芯片检测数据,包括RNA-Seq数据[10]、miRNA芯片数据[11]、CMV数据[12]等大量生物医学信息,与传统医学研究缺乏信息学基础相比,TCGA数据库存储了海量基因组和临床数据供我们分析使用,为基础医学和转化医学构建了桥梁,也为癌症研究及治疗提供了新思路。
参考文献
[1]Juliana Costa-Silva,Douglas Domingues,Fabricio Martins Lopes.RNA-Seq differential expression analysis:An extended review and a software tool[J].PLoS ONE,2012(12):e0190152.
[2]YongKiat Wee,Yining Liu,Jiachun Lu,et al.Identification of novel prognosis-related genes associated with cancer using integrative network analysis[J].Sci Rep,2018,8:3233.
[3]李兆申.胰腺癌早期诊断研究的现状与展望[J].解放军医学杂,2009,34(1):7-9.
[4]黄帅.Bit1蛋白对胰腺癌恶性表型调控作用及机制研究[D].北京:北京协和医院,2016:1-1.
[5]Yuan-Yuan Zhang,Hao-Miao Feng.MEG3 Suppresses Human Pancreatic Neuroendocrine Tumor Cells Growth and Metastasis by Down-Regulation of Mir-183[J].Cell Physiol Biochem,2017,44:345-356.
[6]刘卜玮,蔡明成,杨雪,等.microRNA调控细胞凋亡的研究进展[J].生理科学进展,2018,49(4):309-314.
[7]Shuai Yin,Tim Bleul,Yifan Zhu,Orkhan Isayeva,Jens Werner,Alexandr V.Bazhin.MiRNAs are Unlikely to be Involved in Retinoid Receptor Gene Regulation in Pancreatic Cancer Cells[J].Cell Physiol Biochem,2017,44:644-656.
[8]Zheng Dang,Wei-Hua Xu,Peng Lu,et al.MicroRNA-135a inhibits cell proliferation by targeting Bmi1 in pancreatic ductal adenocarcinoma[J].International Journal of Biological Sciences,2014,10(7):733-745.
[9]Zheng Dang,Wei-Hua Xu,Peng Lu,et al.MicroRNA-135a inhibits cell proliferation by targeting Bmi1 in pancreatic ductal adenocarcinoma[J].International Journal of Biological Sciences,2014,10(7):733-745.
[10]Li Peng,Xiu Wu Bian,Di Kang Li,et al.Large-scale RNA-Seq Transcriptome Analysis of 4043 Cancers and 548 Normal Tissue Controls across 12 TCGA Cancer Types[J].Sci Rep,2015,5:13413.
[11]Gebhardt ML,Mer AS,Andrade-Navarro MA.mBISON:Finding miRNA target over-representation in gene lists from ChIP-sequencing data[J].BMC Res Notes,2015,16(8):157.
[12]Salyakina D,Tsinoremas NF.Viral expression associated with gastrointestinal adenocarcinomas in TCGA high-throughput sequencing data[J].Hum Genomics,2013,27(7):23.
关注SCI论文创作发表,寻求SCI论文修改润色、SCI论文代发表等服务支撑,请锁定SCI论文网! 文章出自SCI论文网转载请注明出处:https://www.lunwensci.com/yixuelunwen/20436.html