SCI论文(www.lunwensci.com):
摘 要:分子叠合是指在三维空间上将两个或多个分子的结构进行重叠,进而比较它们在三维结构和空间性质上差异的操作或方法,在药物、材料等领域的合理分子设计中有广泛的应用。分子叠合的原理和方法属于微观层次, 较为抽象。为了便于分子叠合相关内容的教学,本论文通过模型构建和虚拟现实技术设计了一款分子叠合教学软件。该软件在虚拟课堂中实现语音讲解、三维动画展示、交互式实验操作等功能,生动地讲解了分子叠合的原理、方法分类、相关的软件和应用,使分子叠合中微观、抽象的概念得以直观展现,达到辅助教学的目的。
关键词:分子叠合;虚拟现实;均方根距离;分子场;药物设计
本文引用格式:温聪聪,肖遥,王栋,等 . 基于虚拟现实的分子叠合教学软件 [J]. 教育现代化 ,2021,8(40):10-15.
Virtual reality Course-ware for molecular Superposition TeachingWEN Congcong, XIAO Yao, WANG Dong, KONG Dexin(College of Informatics, Huazhong Agricultural University, Wuhan Hubei)
Abstract: Molecular superposition, which refers to the operation or protocol that overlap the 3D structures of two or more molecules, is extensively applied in the rational design of pharmaceuticals, materials and other functional molecules. The stereostructure or spatial properties of the molecules can be compared with each other. The principles and methods of molecular superposition belong to the microscopic level and are relatively difficult to understand. In order to facilitate the teaching of molecular superposition, in this paper, a course-ware was designed through 3D model construction and virtual reality technology. The software uses virtual classrooms to conduct slides demonstration, audio-visual explanations, 3D animation presentations, and interactive manual experimental operations. The principles and methods of molecular superposition are explained in detail, so that the microscopic concepts in molecular superposition are visually displayed, and the purpose of assisting teaching is achieved.
Keywords: molecular superposition; virtual reality; root mean square deviation; molecular field; drug design
一 引言
分子叠合是一种从三维结构上直接比较分子的方法,它通过在空间上重叠两个或者多个分子,分析它们的空间位置、基团取向关系,为药物、农药、催化剂或有机材料等分子的合理设计提供指导 [1]。但是分子叠合属于微观分子层次的概念,其原理和方法具有高度的抽象性,难以直观理解。虽然教学中可以使用大量的图片或动画,但是这些平面媒体难以呈现分子空间结构的立体感。
虚拟现实技术 (Virtual Reality, VR) 通过计算机生成模拟环境,具有沉浸性、多感知性和互动性等特点 [2],使用该技术可以使学生在虚拟环境中得到逼真的感受和直观的理解 [3],完成一些危险、微观、抽象的实验模拟、见习和理论教学。近年, 虚拟现实技术在化学信息学和计算机辅助药物设计等领域的应用日趋成熟。例如,Baaden 等 [4] 首次实现了 Unity3D 引擎中化学分子结构的可视化。Reymond 课 题 组 [5] 开 发 了 Virtual Reality Chemical Space 软件,使用虚拟现实技术对化学空间进行直观的展示。
Schneebeli 课题组 [6] 将力场参数引入虚拟现实中,从而实现虚拟场景中的分子动力学模拟。然而,上述研究主要集中于在虚拟场景中分子结构的可视化以及物理模型在虚拟现实中的实现,目前还没有关于计算机辅助药物设计方法的虚拟现实教学软件的开发或应用。
为了解决分子叠合教学过程中概念抽象、难以直观展示的问题,本论文基于三维建模和虚拟现实技术,设计了一款分子叠合的教学软件。在软件中采用虚拟现实的技术将分子叠合的概念、方法分类、应用领域和常用软件等知识点以直观的三维模型展示出来。同时,利用虚拟的场景,配合视频的讲解, 结合实验操作,通过视觉、听觉和模拟操作等多感官的人机交互,使分子叠合中晦涩难懂、语言较难解释清楚的理论知识变得容易理解和掌握 [7]。
二 材料与方法
(一) 软件、硬件环境
软件开发过程中使用的三维模型均采用 Blender 2.79[8] 构建,虚拟场景的搭建、VR 实验操作的设计是在 Unity3D 2017.4[9] 中进行的,同时借助 VRTK 3.2.1[10] 以及 SteamVR 1.2.3[11] 两款工具包实现 VR 场景的可视化。分子结构的处理采用的是 MOE 2018 和 VMD 1.9.3 这两款软件。MOE 是一个综合分子操作平台,它可以生成分子的立体结构并将分子进行预处理 [12];VMD 是一款主要用于分子动力学轨迹分析的分子建模软件 [13],它可以将 MOE 预处理好的分子进行渲染并保存为 Unity3D 和 Blender 可以识别的 OBJ 格式文件。
软件开发的硬件环境是惠普图形工作站 Z240, 设备的具体参数为 CPU: Intel®i7-7700; 内存 : 16G RAM;GPU: NVIDIA GeForce GTX1080。开发采用的虚拟现实设备是三星玄龙 MR 头盔,型号 XE800ZAA- HC1。
(二) 技术实现
首先, 使用 MOE 在 Amber 12ETH 力场下对蛋白质和配体复合物的结构进行预处理,确定 pH=7 时的质子化状态,删除与复合物无氢键作用的溶剂分子并进行能量优化。导出预处理过后的结构并保存为 PDB 格式。将预处理后的蛋白质导入到 VMD 中,并用飘带模型显示蛋白质的二级结构。以配体小分子周围 5 埃的氨基酸残基为蛋白质的结合位点, 将蛋白质和配体的复合物模型导出为 OBJ 格式。将OBJ 格式的复合物模型导入 Blender 中,分离出其中的蛋白质模型和配体模型。最后,导入 Unity 中进行渲染和绑定各个对象的控制脚本。
对于待叠合的其它小分子,分别使用 MOE 导出复合物中小分子的三维结构,加氢、加电荷之后进行能量最小化,导出处理好的小分子并保存为PDB 格式。将 PDB 格式的小分子导入 VMD 中,采用 CPK 模型显示小分子并修改小分子 CPK 模型的颜色显示。将经过预处理的小分子保存为 OBJ 格式, 最终将该模型导入 Unity3D 的素材库中。
交互设计是软件设计的一项重要工作,给需要交互的蛋白质和小分子添加 VRTK 的系列控制脚本以及刚体绑定,便于使用手柄对蛋白质和小分子进行操作。通过 VRTK 的移动模块设计脚本,实现人物在虚拟场景中的走动。同时,采用脚本控制手柄射线,设置射线蓝色为可移动区域,红色为不可移动区域。
软件中使用多个羟甲基戊二酰辅酶 A 还原酶(HMGR) 蛋白 - 配体复合物结构来设计叠合动画,以此显示分子叠合的基本概念。采用 C# 语言编写动画脚本,控制蛋白质及配体小分子的移动、显示或隐藏, 从而模拟 HMGR 蛋白或其配基在软件中叠合的过程。为了表征分子叠合结果的优劣,我们分别从待叠合分子与模板分子中选取了四个对应原子,以计算它们的均方根距离 (Root-Mean-Square Distance, RMSD)。RMSD 常用来衡量一个分子或者一个蛋白质与其理想构象之间的差异,其计算如公式(1)所示。
在分子叠合中,RMSD 反映了两个分子叠合结果的优度。RMSD 的值越小, 则说明该分子或蛋白质的构象越接近实验结果。在软件的动画中,我们高亮显示了选定的配对原子及配对原子之间的连接线。在待叠合分子向模板分子移动的过程中,实时显示配对原子之间的距离,将该距离发送到后台以计算 RMSD 值,并实时反馈在虚拟的屏幕上。此外, 为了方便同学们查阅相关专业名词的概念,我们在软件中虚拟屏幕上出现专业名词的位置设计了隐藏触发按钮,学生通过手柄射线指向并单击该按钮就可以控制名词解释的出现和隐藏,方便随时查看。
三 结果与讨论
(一) 教学目标
本软件主要配合理论教学讲解、演示分子叠合的概念、前提假设、方法及分类、应用领域、常用的分子叠合软件等教学内容,简述如下。
分子叠合是计算机辅助药物设计中一种重要的方法,它常用于基于配体的虚拟筛选、药效基团模型的建立、三维定量构效关系分析、药物作用机制的合理预测等方面 [14]。分子叠合的前提假设是所有待叠合的分子与靶标都具有类似的作用模式,即待叠合的分子能够以相同的模式作用于同一个靶标的同一活性位点。通过分子叠合模拟小分子与靶标结合时小分子的构象、位置和取向,可以使分子间的比较更为直观、有意义。
按照叠合的目标设定,分子叠合可以分为基于模板的叠合和一致性叠合;按照叠合过程中是否考虑小分子的柔性 ( 构象变化 ),还可以分为刚性叠合和柔性叠合。分子叠合的过程包括叠合和打分,其中叠合过程主要是完成分子构象的采样 ( 柔性叠合 ) 和叠合模式 ( 位置取向 ) 的遍历,一些软件也将构象产生和叠合过程同时进行;而打分主要是用于分子叠合优度的评价。如何控制分子叠合的算法复杂度以及提高叠合打分与分子活性之间的相关性是分子叠合算法开发的难点。
常用的分子叠合方法主要分为四种:1) 基于原子对最小 RMSD 的叠合,这是最简单但最重要的分子叠合方式。采用这种方式进行叠合时,通常将分子视作刚性,如有必要,可先进行构象空间采样。目前,蛋白质之间的结构叠合就主要采用基于 Cα 原子或主链原子 RMSD 最小化的叠合方式 [15];2) 基于分子场的叠合,是基于分子对周围空间呈现出的分子力场特性而进行分子间的柔性叠合。通常分子的立体因素和电性因素各占最后打分评价的 50%。这种方法的叠合速度较慢,但结果具有较为明确的生物学意义 [16];3) 基于惯性矩、偶极矩的叠合,主要是适用于分子对接中,将小分子放入蛋白质的活性腔中以快速获取分子的初始位置和取向。这种叠合方式的叠合速度较快,但结果并不准确;4) 基于受体的叠合,将多个分子分别与靶标蛋白进行分子对接,就获得了这些分子的空间叠合关系。这种叠合方法能够很好反映药物与靶标的分子间相互作用, 但是叠合的结果受到对接打分函数的准确度和空间位阻基团的影响 [17]。
(二) 分子叠合教学设计
为了实现以上教学目标,本教学软件以分子叠合的概念为切入点,讲解了分子叠合的前提、目的、难点以及重要性等基础知识;然后,把分子叠合的四种方法 ( 即基于原子对最小 RMSD 的叠合、基于分子场的叠合、基于惯性矩 / 偶极矩的叠合和基于受体的叠合 ) 做了详细的讲解以及动画演示; 接着, 介绍了分子叠合的应用场景以及目前主流的分子叠合软件,包括 FlexX、ROCS、MOE、Surflex-sim 等; 最后对分子叠合虚拟课程的内容做了整体回顾。在课程结束还设置了课后实验操作, 帮助学生巩固学习到的知识。软件学习的整体流程如图 1 所示。
(三) VR 场景设计
图 1 分子叠合虚拟仿真教学的课程设计了学生有一个较好的学习收获,课程开始到课程结软件场景设计在 Unity3D 引擎中完成,充分利用 Unity3D 跨平台开发的优势,使学生能够在虚拟环境中获得真实的实验体验 [18]。分子叠合教学软件共包括 3 个场景:初始场景、跳转场景、教学主场景。其中,初始场景和跳转场景与本课题组已经开发完成的其它系列虚拟现实课程共用。
而教学主场景是教学的主要环境,包括一个虚拟的教室以及其中的计算机和操作台等三维模型,尽可能真实地还原教室环境。在这个场景中可以完成分子叠合虚拟课程的演示教学和所有虚拟实验操作。在虚拟课程中为束的整个过程中设定人物不能随意移动。在课程结束后,学生通过 VR 手柄操作,可在虚拟教室自由参观,观看实验室的墙报、虚拟物体 ( 细胞模型、药物分子 )。虚拟教室的左侧试验台上放置一系列的小分子和蛋白质,可供学生在课程结束后拿起观察、使用。整个虚拟教室场景如图 2 所示。
(四) 基于 VR 的分子叠合实验
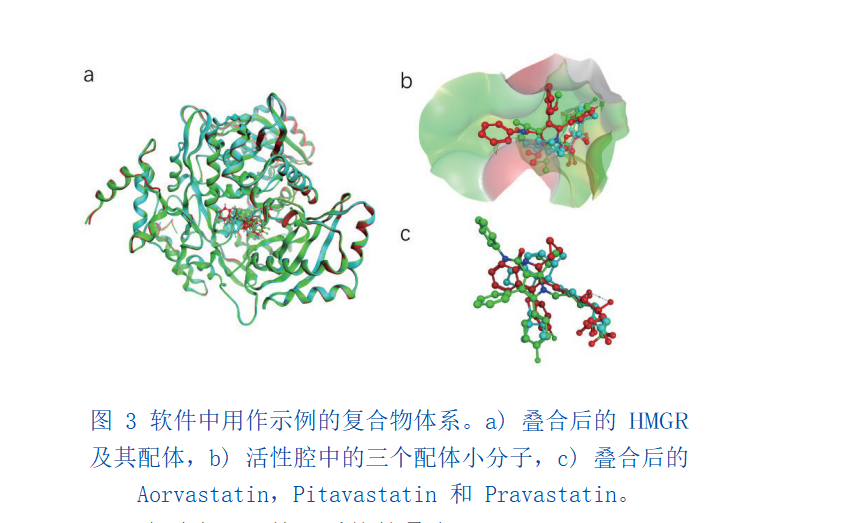
为了加深对分子叠合的理解,我们以 HMGR 及其抑制剂的晶体复合物的体系为例设计了一个动画和三个互动实验。HMG-CoA 还原酶 (HMGR) 是细胞质固醇类化合物代谢中的重要调控点,是降血脂药物设计的重要靶标,抑制该酶的活性可以有效地降低血浆总胆固醇水平,从而降低心脑血管疾病的发病几率 [19];而他汀类药物作为 HMGR 的抑制剂是目前最有效的降血脂药物, 例如 Aorvastatin、Pitavastatin 和 Pravastatin 等。在本软件中以 HMGR 和上述三种抑制剂为示例体系进行虚拟实验设计 [20]。HMGR 及其抑制剂的晶体复合物结构如图 3 所示。
动画演示:基于受体的叠合在讲解了分子叠合的概念以后,软件会展示三个HMGR 及其抑制剂的复合物结构,每个复合物中包含一个不同的HMGR 抑制剂。然后,以动画的形式演示受体结构的叠合过程。在这个体系中,三个配体小分子在 HMGR 蛋白的活性腔中结合模式相同 / 相似。蛋白质叠合之后,可以获得小分子的结构关系,使学生们理解分子叠合的前提、目的和意义。
演示过程中, 会根据教学需要动态隐藏蛋白质飘带、活性腔的表面模型。最后,将叠合好的配体小分子移动到学生面前, 学生可以利用手柄拿起叠合后的配体小分子进行观察和比较。图 4a 展示了待叠合的三个HMGR 及其配体的复合物,图 4b 为拿起并观察叠合后小分子结构的情形。
互动实验:基于原子对最小 RMSD 的叠合为了更清晰地讲解基于原子对最小 RMSD 的叠 合 , 教 学 软 件 中 以 Atorvastatin 为 模 板 分 子 , Pitavastatin 和 Pravastatin 为待叠合分子,分别设置了相同分子、相同构象之间的叠合、相同分子不同构象之间的叠合以及不同分子之间的叠合三种虚拟实验操作,并以动画形式展示这三种情况的叠合过程。使用此种叠合方法进行分子叠合时需要在分子间选择配对原子,在软件中设置将配对原子高亮显示,同时显示它们之间的连线、距离和 RMSD 值。
将在分子模拟软件 ( 如 MOE) 中迅速完成的操作,一步一步拆解开演示给学生,帮助他们更好地理解分子叠合的方法。在动画结束之后,还设置了三种不同情况下的 RMSD 叠合的 VR 实验操作。同样使用动画中的三种他汀类小分子作为实验素材,以 Atorvastatin 为模板,其余三个他汀类小分子 ( 包含 Atovastatin 本身 ) 为待叠合分子,学生可通过 VR 手柄拿起待叠合分子,与模板分子进行叠合。在实验操作过程中需要学生挑选配对原子,软件根据学生的选择确定不同的叠合情况,实时给出 RMSD 值,帮助学生判断叠合优劣。
在达到最佳叠合之后,模板分子与待叠合的小分子之间的分子标签同样重合在一起。图 4c 为 Atorvastatin 和Pitavastatin 的叠合过程截图,展示了配对原子之间连线、距离和实时计算 RMSD 值反馈到虚拟屏幕中的情形。图 4d 为在软件中进行基于原子对最小 RMSD
四 总结与展望
分子叠合是一种十分重要的药物设计方法,是基于配体形状虚拟筛选和三维定量构效关系研究等方法的基础。在实际分析中,分子叠合过程是使用软件自动完成的,其背后的原理和方法常常由于难以直接认知而被忽略。
本文报导了一款基于虚拟现实技术的分子叠合教学软件,通过介绍叠合基本概念、讲解叠合原理方法、模拟叠合整体过程,将晦涩难懂的分子叠合概念直观地展示到学生面前,他们可以边听边自己动手操作,增加了课堂的趣味性,提高了知识的传播效率。
近年来,各高校在实验教学上广泛使用了虚拟仿真技术 [21],方便学生熟悉实验的条件和过程,避免潜在的危险或昂贵实验材料的浪费。与这些教学软件大多借助键盘或鼠标进行交互不同,本文实现的基于虚拟现实的分子叠合教学软件通过 VR 穿戴设备实现,不仅可以在虚拟空间中查看分子叠合的过程,还可以通过 VR 手柄拿起分子,进行叠合操作,大大提高了实验过程的真实感和教学效果。
与传统的虚拟实验相比,沉浸式 VR 设备在教学中也有一些弊端。例如,长时间佩戴 VR 头盔会引起一定的眩晕感。本软件在设计时,将教学时间控制在15-20 分钟以内,避免产生不适。此外,本软件需要配合头盔、手柄等虚拟现实设备使用,疫情期间学生无法到校学习,所以基于 VR 的教学活动无法在线开展。
目前,本课题组已经完成了基于虚拟现实的分子对接、分子叠合和同源模建三个教学软件的开发。这些 VR 教学软件在多门本科生或研究生课程 (《药物设计导论》《分子模拟与药物设计》等 ) 中试用, 学生们的积极性、参与度和教学效果有很大的提升。同时, 作为生物过程虚拟仿真实验室的入学教育、社会服务展示项目,在我校 120 周年校庆、对外科普教育等活动中发挥了巨大的作用,得到了社会各界的一致好评。下一步,本课题组还将继续开发《计算机辅助药物设计》课程中其它概念或方法 ( 如三维定量构效关系、多片段搜寻等 ) 的 VR 教学软件。
参考文献
[1]胡奔 . 三维生物相关谱在计算机辅助药物设计中的应用 [D]. 武汉 : 华中农业大学 , 2017.
[2]陈雪梅,杜棋东 . 基于虚拟现实技术的交互式微课设计研究[J]. 教育现代化 ,2019,6(46):279-281.
[3]Dyer E, Swartzlander B.J, Gugliucci M.R. Using virtual reality in medical education to teach empathy [J]. Journal of the Medical Library Association : JMLA 2018, 106(4): 498-500.
[4]Perez S, Tubiana T, Imberty A, Baaden M. Three-dimensional representations of complex carbohydrates and polysaccharides- SweetUnityMol: A video game-based computer graphic software [J]. Glycobiology, 2014, 25(5): 483-491.
关注SCI论文创作发表,寻求SCI论文修改润色、SCI论文代发表等服务支撑,请锁定SCI论文网!
文章出自SCI论文网转载请注明出处:https://www.lunwensci.com/jiaoyulunwen/32409.html