SCI论文(www.lunwensci.com)
摘 要: 目前,国内外已有许多动物基因组学数据库,却还未有专门针对蒙古高原家畜基因组信息构建的数据库。此外, 传统的基因组数据库平台一般采用关系型数据库存储数据,但在面对海量的基因组数据时出现了读写性能差、可靠性低、不易 扩展等问题。为解决上述问题,收集整合了牛、绵羊、山羊、骆驼等蒙古高原家畜的基因组数据,应用非关系型数据库,设计 并实现了基于 MongoDB 存储架构的蒙古高原家畜基因组大数据管理系统。该系统的实现为蒙古高原家畜分子生物学研究提供 了一个良好的数据平台,也解决了海量基因组数据的存储与管理问题。
关键词:基因组学数据库 ;蒙古高原家畜 ;海量数据 ;非关系型数据库 ;MongoDB
Design and Implementation of Mongolia Plateau Genome Big Data Management System Based on MongoDB
WU Xuemin1. GAO Jing2
(1.Department of Computer and Information Engineering, Baotou Vocational and Technical College, Baotou Inner Mongolia 014000;2.College of Computer and Information Engineering, Inner Mongolia Agricultural University,Hohhot Inner Mongolia 010000)
【Abstract】: At present, there are many animal genomics databases at home and abroad, but there is no database specially built for the genome information of livestock on the mongolian plateau. In addition, the traditional genome database platform generally uses relational databases to store data, but in the face of massive genome data, problems such as poor read-write performance, low reliability, and difficult expansion have emerged, In order to solve the above problems, we collected and integrated the genome data of cattle, sheep, goats, camels and other livestock on the mongolian plateau, and designed and implemented a big data storage system for the genome of livestock on the mongolian plateau based on the MongoDB storage architecture using a non relational database. The implementation of this system provides a good data platform for the molecular biology research of livestock on the Mongolian plateau, and also solves the problem of storage and management of massive genome data.
【Key words】: genomics database;mongolian plateau livestock;massive data;non-relational database;MongoDB
引言
随着测序技术的持续发展以及实验数据的不断积 累,当前已形成大量一级生物信息数据库,如 NCBI、 ENSEMBL 及 USCS。它们各自按一定的目标归类整合实验的原始数据,并提供数据查询、下载及处理等服 务。但是在实际研究中,一级数据库不一定能满足研究 人员特定的数据及功能需求,因此,针对不同研究内容 及需要,进一步整合一级数据库中的知识和信息,通过系统化创建专用二级数据库。
当前,关于动物基因组学的二级数据库非常多,如 牦牛基因组数据库、家蚕基因组数据库和猪基因组数据 库 [1-3],却大多集中在全球普遍饲养的动物,还未构建 专门针对蒙古高原家畜研究的基因组数据库。此外,随 着生物科学技术的飞速发展,蒙古高原家畜基因组数据 量已增长到 TB 级别,基于关系型数据库的传统存储方 式在应对大数据时显露出不足。
本文收集整合了牛、羊、骆驼等蒙古高原家畜的基因 组数据,通过分析数据的特征,制定了可行的 MongoDB 存储架构,并采用业界当前的主流技术,实现了蒙古高 原家畜基因组大数据管理系统,测试结果表明该系统具 有良好的读写性能。
1 蒙古高原家畜基因组数据的特征及传统存储方式的问题
为了给蒙古高原家畜基因组大数据管理系统的构建 提供充分的数据支持,目前已从 ENSEMBL 和 NCBI 等公共数据库下载整合了羊、牛、骆驼等蒙古高原家畜 的基因组数据 17000 多条,共约 3TB 的数据量,通过 分析已有的数据资源,总结了蒙古高原家畜基因组数据 具有以下几个特征 :
(1) 结构复杂。除了有结构化的描述信息,还有非 结构化的序列文件,如 .sra 文件 ( 高通量测序数据序列 文件 ) 以及 .gbff 文件 ( 全基因组序列文件 )。
(2)格式多样。由于数据的来源众多,格式不一, 所以不同序列文件所需存储的描述信息不尽相同。
(3)大文件。单个序列文件达 GB 级别。
(4)数据量大。目前数据总量已达到 TB 级别。
此外,蒙古高原家畜基因组数据在实际业务工作中 还具有以下特征 :
(1)集中写入大批量数据的业务较多,导入之后, 更改操作很少发生。
(2) 需支持多种查询方式,并且具有良好的查询性能。
(3)随着测序技术的发展,数据量还在不断增加, 要求数据库具有很好的可扩展性。
综上所述,蒙古高原家畜基因组数据呈现出海量、 大文件、结构复杂等一系列大数据特征。
传统的基因组数据存储系统都是基于关系型数据库 设计研发的,提供给数据存储和管理一个高效率的平台, 但由于自身的限制,在面对蒙古高原家畜基因组大数据 时存在着许多的弊端。首先,关系型数据库需要预先设 计固定的表结构,因此无法灵活添加多样化的基因组信 息 ;其次,因为 GB 级别的序列文件不断增加,造成关 系型数据库的体积持续增大,读写性能不断下降 ;最后,关系型数据库集群架构存在着关系复杂、维护成本高、 代码处理复杂及难以横向扩展的问题。然而 NoSQL 非 关系型数据库恰恰能够弥补以上所列举的不足之处。其 中, MongoDB 数据库发展较好,以其高性能、易部署、 高可用的优点成为了关系型数据库的替代品 [4]。
2 基于 MongoDB 的存储架构
MongoDB 由 C++ 语言开发,是一款面向文档的 分布式数据库,为海量大数据的存储问题提供了高性 能的解决方案。再者, MongoDB 面向集合存储,采用 BSON 作为基本存储单位,并且模式自由,因此可根据 实际需求动态地插入格式多样的基因组序列相关描述信 息。另外, MongoDB 具有丰富的查询表达方式,完全 能够满足蒙古高原家畜基因组数据的查询需求 [5.6]。
分布式文件系统 GridFS 是 MongoDB 的一个子模块, 提供了大型二进制数据在数据库中的解决方案,可用来存 储大型基因组序列文件并且支持分布式应用。GridFS 将 巨大文件分割成很多小文件块并以两种集合配合的方法 存储文件。Files 集合来存储文件相关信息, Chunks 集合用来存储二进制文件块,两集合用 ID 对应。默认 情况下, MongoDB 用时间戳 + 机器 + 进程标识 + 计 算器计算获得 ID,它具有高度的唯一性,可达到秒级。 另外, GridFS 通过复制和自动分片机制突破了一般文 件系统对文件的限制,故障恢复能力强,扩展性好 [7]。
2.1 存储方案
前文提及的 MongDB 的文档型存储模式和 GridFS 的大文件存储模式在蒙古高原家畜基因组大数据存储方 面具有很多优势,但也存在一些各自的不足,文档型存 储模式虽然可以有效的存储基因组的描述信息, 但对于序 列文件本身却因其长度远远大于 16M 而无法有效的存储。 相反,基于 GridFS 的大文件存储方式可有效的存储序列 文件,但对于格式多样的描述信息却无能为力。因此本文 设计了一种新的存储方案,如图 1 所示, 将 MongoDB 和 GridFS 有效结合,存储优势得到了更大的发挥。
首先采用 GridFS 的大文件存储方式将序列文件插入 数据库中, 然后利用第三方编程语言, 通过 MongoDB 提 供的与之对应的 API 接口,采用 Excel 批量数据导入 的方式获取有关序列文件的相关描述信息,并将其通 过传统的文档型存储模式以一条文档信息形式插入集合 中。在此过程中最重要的是需要将该序列文件 ID 作为 一条记录存入描述的文档中,以此关联序列文件的 ID 和描述信息的 ID,因此, 外部通过一条文档中记录的 序列文件 ID 就可以快速检索到与该文档记录信息对应 的序列文件并进行读取下载。
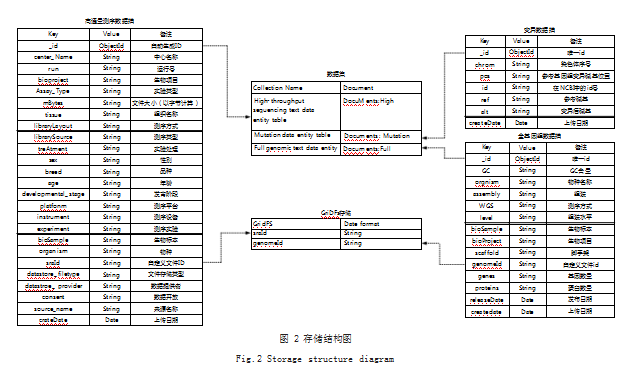
2.2 存储结构
本系统中整合的绵羊、山羊、牛及骆驼基因组数据 有三类,第一类为全基因组数据,可供研究人员对于蒙 古高原家畜完整的基因信息进行分析 ;第二类为高通量测 序数据,可供研究人员对于蒙古高原家畜的血液、肌肉、 胃和肠等器官组织的基因信息进行分析 ;第三类为变异 数据,可供研究人员对目前已知的变异情况进行了解。
本文结合 MongoDB 的存储方案和基因组数据的分 类信息,构建了两层的数据库存储结构,分别为元数据 层和具体的数据集。用户通过元数据了解数据库中数据集的描述信息,并根据元数据提供的数据类型进行读取 和转换数据格式。
(1)元数据,元数据集中包含蒙古高原家畜基因组 数据集的内容描述以及每项数据的类型和含义。
(2)数据集,建了三个集合来存储三类数据的描述 信息,分别为全基因组数据集、高通量测序数据集及变 异数据集,如图 2 所示。
高通量测序数据集中文档由自动生成 ID(_id)、自定 义文件 ID、运行号(Run)、生物项目(Bioproject)、 实验类型(Assay_Type)、文件大小(mBytes)、组织 名称(Tissue)、测序方式(Library Layout)、测序类型 (Library Source)、实验处理(Treatment)、性别(Sex)、 品种(Breed)、年龄(Age)、发育阶段(developmental_ stage)、测序平台(Platform)、测序设备(Instrument)、 测序实验(Experiment)、生物标本(bioSample)、物 种(Organism)、中心名称(center_Name)、文件存储类型 (datastore_filetype)、数据提供者(datastroe_provider)、 数据开放(Consent)、来源名称(source_name) 及上传日 期(CrateDate) 组成。其中序列文件存储在 GridFS 中, 文档中只记录序列文件的 ID。全基因组数据及变异数据 的文档结构与高通量测序数据存储结构相似,不再赘述。
2.3 分布式集群方案
目前单台服务器已无法满足如此大的蒙古高原家畜 基因组数据量。为了解决了海量数据的存储问题,备份数据,本文将 MongoDB 数据库设计成集群模式,搭载 多台服务器,一同工作,来应对大数据带来的挑战。
MongoDB 的服务器节点由多种类型,集群支持多种 部署方式 :如复制集群、分片集群、分片 + 复制集群 [8]。 分片可用于缓解高吞吐数据的负载压力过大、数据延迟 和内存过载等情况 ;当发生故障时,复制集可保证数据 的完整性和整个服务器的正常工作 [9.10]。于是为了分散 各节点的负载,保持数据的同步,保证整个服务器环境 的持续运行,本文采用分片和复制集群结合的方式存 储数据,设计方案如图 3 所示。MongoDB 集群目前由 三台服务器组成, 其中包含三个路由服务(Mongos)、 三个配置服务(Config)、三组数据分片(Shard)。
由于路由服务器和配置服务器不需要存储大量数 据,因此将其置于数据分片所在的服务器上,从而充分 利用硬件资源。集群中的分片存储整个蒙古高原家畜基 因组数据中的一部分,随着数据的增长,增加额外的片 可以增加集群的存储能力。每一个副本集包可以含 1 个 主节点、1 个从节点和 1 个仲裁节点,写入主节点的数 据被异步同步到从结点,实现最终一致性。当主节点出 现故障的时候,不需要人工干预的情况下仲裁节点就会 迅速采取相应措施将从节点提升为主节点,此策略保证 了数据的高可用性,实现了自动故障转移。另外,将主 节点与副节点以及仲裁节点放在不同的服务器上,从而 保证数据的可靠性和安全性。
3 系统设计
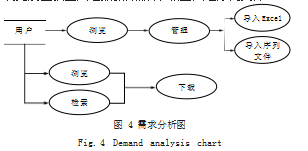
3.1 数据库需求分析
数据库的需求分析主要结合系统的功能分析,如 图 4 所示。可以将操作过程分成两部分 :第一部分用户可以浏览及管理本地的基因组数据,通过 Excel 文 件将基因组数据进行批量导入,也可将序列文件导入 MongoDB 数据库 ;另一部分就是数据处理,用户可以 浏览及查找基因组数据,然后下载基因组序列文件。
3.2 总体架构设计
本系统底层将服务器, 存储、网络整合成一个虚拟 化资源池,可根据应用系统的需要灵活分配及调动物理 资源,提高了资源利用效率,再加上资源池动态可伸缩 的特性,非常适用于分布式系统的部署。数据层采用 MongoDB+GridFS 分布式集群,保障了整体功能的负 载均衡与故障恢复。接口层对外提供统一的数据访问接 口、文件按访问接口及查询接口,使上层应用与数据层 之间的交互流程简单化。展示层为用户提供了友好的界 面,操作简单。设计方案如图 5 所示。
4 系统开发与测试
4.1 应用运行环境
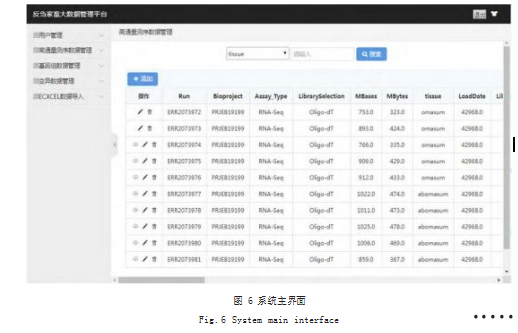
蒙古高原家畜基因组大数据管理系统基于 C/S 架构, 采用 HTML、JavaScript 及 VUE 框架等 Web 技术构建 用户界面。后端基于 Spring MVC 架构,使用 MongoDB 分布式集群,采用 Java 语言进行开发。如图 6 所示,用户可在 Web 界面执行管理、检索、下载等操作。
4.2 存储平台部署
本文采用 3 台服务器组建 MongoDB 分布式集群,每 台服务器配置相同,服务器配置如下 :CPU 为 4 核、内 存容量 8GB、磁盘容量 1TB, 操作系统为 Ubuntu 20.04. MongoDB 版本为 3.6.前文已介绍过采用的分片复制 集群架构,具体配置信息如表 1 所示。
4.3 系统测试
实验验证该存储平台的读写性能。首先对 130 万多 条文档数据写入 MongoDB 分布式集群,与同等条件下 SQL Server 2012 内同等数据量写入时间进行对比。然后对 130 万条基础数据量的 MongoDB 分布式集群内单条 数据查询时间, 与同等条件下 SQL Server 2012 内单条数 据查询时间进行对比。如表 2 所示显示了读写性能测试对 比结果,可以看到基于 MongoDB 分布式集群的蒙古高原家 畜基因组大数据管理系统提供了良好的读写性能。
5 结语
本文设计并实现了基于 MongoDB 存储架构的蒙 古高原家畜基因组大数据管理系统。该系统比采用关系 型数据库的传统系统具有更好的性能,提供给用户高性 能、无模式和自动扩展的数据存储。另外,该系统的构 建能够为从事蒙古高原家畜研究的科学工作者提供方便 和帮助,并能够为相关数据库的构建积累经验。
参考文献
[1] 黄廷华,曹建华,余梅,等.猪专门化分子生物学数据库的建立 及其初步应用[J].猪业科学,2006.23(2):19-20.
[2] 张文广,陈铭,李金泉.绒山羊分子生物学数据库设计与构建 [J].中国生物工程杂志,2005(4):199-203.
[3] 胡泉军.牦牛基因组数据库建设[D].兰州:兰州大学,2014.
[4] 赵永强.基于NoSQL的特色数据库系统研究[J].图书馆工作 与研究,2018(S1):97-99+124.
[5] 徐旭平,李小勇.基于MongoDB 的元数数据管理理研究[J]. 信息技术,2018.25(8): 95-101.
[6] 郭远危.大数据存储MongoDB实战指南[M].北京:人民邮电 出版社,2015.
[7] 邱新忠.基于MongoDB GridFS的地图瓦片数据存储研究 [J].地理空间信息,2016.17(2):50-52.
[8] 杨宏章.MongoDB分布集群方案部署[J].中传媒科技,2021. 31(3):111-113.
[9] 刘仪轩.基于MongoDB的排水设施监管养护系统的应用与 研究[D].上海:东华大学,2020.
[10] 符永琦.基于MongoDB的高可用性分布式数据库集群技 术研究[J].信息技术与信息化,2020.36(9):56-58.
关注SCI论文创作发表,寻求SCI论文修改润色、SCI论文代发表等服务支撑,请锁定SCI论文网!
文章出自SCI论文网转载请注明出处:https://www.lunwensci.com/jisuanjilunwen/53642.html